D. LA SURVEILLANCE VIA LES EAUX USÉES
1. L'intérêt de l'épidémiologie à partir des eaux usées
Les eaux usées sont le réceptacle des matières fécales et des fluides corporels excrétés par les humains. Elles contiennent ainsi une large variété de contaminants, comme des métabolites de médicaments ou des agents pathogènes (bactérie, virus, parasite), et leur analyse permet de mener diverses études épidémiologiques.
Le suivi des eaux usées présente plusieurs avantages significatifs en termes de suivi des maladies infectieuses. Tout d'abord, il fournit des résultats indépendamment de l'état de santé des individus, que ceux-ci soient symptomatiques ou asymptomatiques, tant qu'ils excrètent une quantité de matériel viral suffisamment importante pour que celui-ci puisse être caractérisé. Il permet de suivre l'ensemble d'une population sans être influencé par la stratégie de dépistage mise en oeuvre, l'efficacité de celle-ci dépendant de la sensibilité et de la spécificité des tests, variables selon la maladie, de leur disponibilité, qui peut être limitée, et du recours à ceux-ci, susceptible d'induire des biais. Il ne dépend pas non plus de l'accès à l'offre de soins, également susceptible de souffrir de limitations, comme les indicateurs issus de la médecine de ville ou hospitalière.
Cette méthode permet d'obtenir des informations relativement rapides pour suivre la dynamique d'une maladie infectieuse dans une temporalité proche du réel296(*), contrairement à la plupart des indicateurs traditionnellement utilisés. Cela la rend particulièrement efficace pour détecter précocement l'émergence de nouveaux foyers épidémiques et pour mettre en oeuvre rapidement des mesures de santé publique adaptées (mesures d'atténuation, déploiement d'une stratégie de dépistage, etc.). L'intérêt est donc particulièrement marqué pour les maladies dont les symptômes ne sont pas spécifiques ou tardifs, susceptibles d'entraîner des consultations médicales retardées, et celles pour lesquelles les outils diagnostiques sont peu disponibles ou faiblement efficaces. Enfin, cette approche permet de suivre de vastes groupes de population à des coûts relativement limités par rapport aux méthodes de suivi traditionnelles basées sur des tests de dépistage. En revanche, elle ne fournit qu'un suivi communautaire, qui ne permet pas d'estimer avec précision le nombre de personnes infectées, de localiser les clusters de manière précise297(*) ou de recueillir des informations sur les caractéristiques des personnes contaminées ou sur la gravité de la maladie.
Initialement développée aux États-Unis pour suivre la poliomyélite, cette technique reste aujourd'hui utilisée dans certains pays où cette maladie est endémique298(*) ou, de façon ponctuelle, en cas de résurgence299(*). Cette approche a également été utilisée pour détecter les bactéries du choléra300(*) ou de la fièvre typhoïde301(*), ainsi que plusieurs virus entériques302(*). Dans certains cas, les eaux usées ont également servi à obtenir des informations collectives sur la consommation de drogues, comme dans le cadre du réseau Score (Sewage analysis Core group) de l'Observatoire européen des drogues et des toxicomanies303(*), ou de médicaments, comme les antidépresseurs304(*).
2. L'utilisation dans le cadre de la covid-19
a) Un intérêt confirmé pour le suivi du SARS-CoV-2
Grâce aux connaissances acquises à partir des précédents coronavirus, il a été envisagé dès le début de la crise sanitaire que le SARS-CoV-2 puisse être présent dans le tube digestif et excrété dans les matières fécales, ouvrant la voie à un suivi de l'épidémie par l'intermédiaire des eaux usées. Dans un contexte de capacités de dépistage limitées, cette méthode offrait une formidable opportunité de surveiller la propagation du virus de manière exhaustive, en tenant compte des personnes asymptomatiques et pré-symptomatiques305(*).
Dès le début de l'année 2020, des recherches menées aux Pays-Bas ont mis en évidence la possibilité de quantifier le SARS-CoV-2 dans les eaux usées et l'intérêt de cette méthode pour détecter la circulation du virus avant même que les premiers cas soient déclarés306(*). De manière similaire, en France, le consortium Obépine (Observatoire épidémiologique dans les eaux usées) a montré dès mars 2020 la présence du virus dans les stations d'épuration d'Île-de-France et la possibilité d'observer l'effet du premier confinement sur la réduction de la charge virale307(*). Ces travaux séminaux ont ainsi démontré la faisabilité d'utiliser l'analyse des eaux usées pour suivre l'épidémie, de manière quantitative et dans une temporalité pertinente pour une approche de surveillance épidémiologique.
De multiples travaux académiques conduits alors ont montré la possibilité de construire, à partir du suivi des eaux usées, des indicateurs dont la dynamique est corrélée à la circulation du virus en population humaine et qui permettent d'anticiper les tendances épidémiologiques, afin de fournir une information précoce aux pouvoirs publics, à la population et aux professionnels de santé308(*). Portée par ces résultats encourageants, cette approche a rapidement suscité un vif intérêt à l'échelle mondiale, entraînant la mise en place de nombreuses initiatives309(*). Principalement utilisée dans les stations de traitement des eaux usées, cette méthode a également pu être mise en place à l'échelle d'un bâtiment310(*) ou d'un quartier311(*), permettant une identification plus précise des éventuels foyers de contagion et des situations de reprise épidémique.
Les travaux de recherche menés au cours de la crise ont démontré que les données issues des eaux usées étaient susceptibles d'avoir une avance allant de 0 à 14 jours par rapport à celles issues des tests individuels312(*). Ce délai peut toutefois considérablement varier au sein d'un même réseau de surveillance, selon la station considérée313(*) et en fonction de l'efficacité de la stratégie de dépistage parallèlement mise en place314(*). Il a par exemple été montré dans le Massachusetts que, si le suivi des eaux usées avait précédé les cas cliniques déclarés lors de la première vague de la pandémie, un tel effet n'avait pas été observé au cours de la deuxième vague, en raison de l'augmentation des capacités de dépistage permettant de détecter et de déclarer les cas plus rapidement315(*). De plus, la succession des différents variants est également susceptible d'affecter les paramètres de l'excrétion virale et d'avoir un impact sur la précocité du suivi. À titre d'exemple, en Californie, l'avance du suivi des eaux usées a diminué avec l'apparition du variant Delta et même disparu avec l'émergence du variant Omicron316(*). Enfin, la couverture vaccinale de la population et l'immunité naturelle influent également sur l'excrétion virale, ajoutant ainsi une nouvelle source de variation dans le suivi des eaux usées, tant sur le plan spatial - l'immunisation n'étant pas nécessairement homogène - que temporel.
De manière similaire, le rapport entre les concentrations virales mesurées dans les eaux usées et le nombre de personnes infectées varie, à la fois dans l'espace et dans le temps. Du fait d'un manque de connaissances sur le taux et la dynamique d'excrétion du virus, qui dépendent de nombreux facteurs (âge, sexe, statut immunitaire, variant contracté, gravité de la maladie, etc.), mais du fait également de l'influence potentielle des précipitations, des infiltrations ou des rejets d'eau industriels, qui peuvent induire une certaine dilution317(*), il apparaît aujourd'hui impossible, compte tenu des connaissances disponibles, d'estimer précisément le nombre de personnes infectées en se basant uniquement sur la quantité virale détectée dans les eaux usées. Il en résulte une certaine incertitude qui relativise l'interprétation épidémiologique des indicateurs issus des eaux usées ; un variant associé à une excrétion virale plus élevée - comme cela semble être le cas du récent variant JN.1318(*) - peut ainsi conduire à une surestimation de la reprise épidémique.
En complément du suivi des tendances épidémiques, l'analyse par séquençage des échantillons prélevés dans les eaux usées permet de suivre la diversité génétique virale en circulation. En France, des travaux exploratoires ont montré, dès 2021, la possibilité de suivre les dynamiques de circulation des variants dans la population à partir des eaux usées319(*). Cette approche a l'avantage de pouvoir analyser l'ensemble des variants en circulation et, ainsi, de s'abstraire du biais induit par le recours au dépistage. Cela permet d'observer des variants dits « cryptiques », rarement identifiés parmi les échantillons cliniques320(*). À l'échelle internationale, plusieurs travaux ont montré que cette approche pouvait offrir des résultats comparables à ceux obtenus par l'usage de tests individuels321(*) et permettre de détecter les futurs variants d'intérêt322(*) avec une certaine précocité, comme pour les tendances épidémiologiques.
À l'heure actuelle, alors que le nombre de tests a considérablement diminué, l'intérêt du suivi des eaux usées apparaît, malgré les limites soulignées, plus pertinent que jamais pour surveiller et anticiper la circulation et l'évolution du SARS-CoV-2. De plus, le caractère coût-efficient de cette approche323(*) la rend particulièrement adaptée pour assurer un suivi continu de la population au long terme, contrairement à une stratégie de dépistage à grande échelle324(*).
b) Évolution du dispositif français
Le consortium de recherche Obépine s'est formé dès le début de l'année 2020 autour de virologues, microbiologistes, hydrologues et modélisateurs, afin d'évaluer le potentiel d'un suivi épidémiologique du SARS-CoV-2 via les eaux usées325(*). De premiers travaux encourageants sur l'intérêt de cette méthode ont permis au Comité analyse, recherche et expertise covid-19 (CARE) d'identifier cette stratégie comme un axe de recherche prioritaire. Le consortium Obépine a ainsi pu bénéficier d'un financement de 3,5 millions d'euros du ministère de l'Enseignement supérieur, de la Recherche et de l'Innovation pour mettre en oeuvre un réseau expérimental à l'échelle nationale, afin d'étudier la faisabilité d'une telle surveillance pendant un an. Les recherches ont permis de mettre au point des protocoles d'analyse et d'exploitation des données, d'identifier une stratégie d'échantillonnage et de construire un réseau de laboratoires pour suivre en routine la dynamique de circulation du virus dans plus de 200 stations. Les résultats de ces travaux ont été présentés en accès libre sur un site internet dédié326(*).
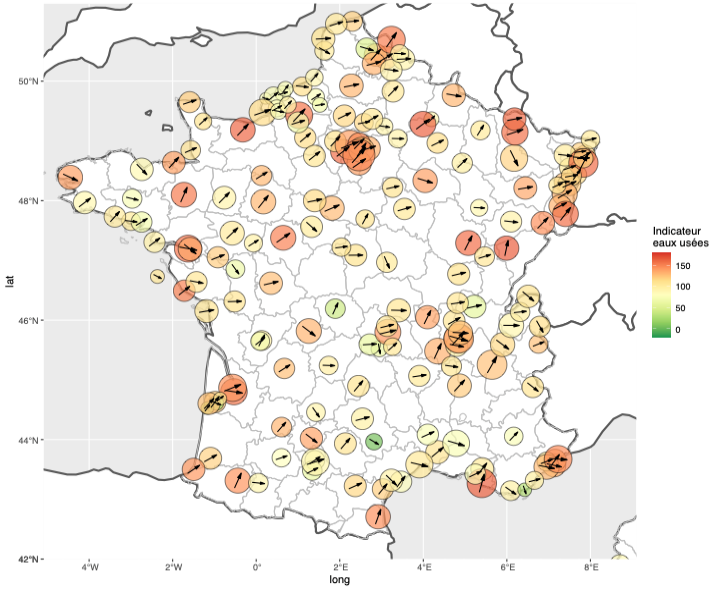
Exemple de synthèse des tendances de l'évolution de l'indicateur Obépine sur 7 jours
Note : La couleur du disque correspond à la valeur de l'indicateur, l'orientation de la flèche à la tendance de l'indicateur et la taille du disque à la taille de la station.
En 2021, à la lumière des résultats des diverses initiatives déployées dans le monde, la Commission européenne a recommandé aux États membres de mettre en place un système national de surveillance des eaux usées pour recueillir des données sur la présence du SARS-CoV-2 et des variants dans les eaux usées327(*). En France, le ministère des Solidarités et de la Santé a annoncé la création d'un dispositif national pérenne de surveillance microbiologique des eaux usées, dénommé SUM'Eau (Surveillance microbiologique des eaux usées), sous le pilotage de la Direction générale de la santé et la Direction de l'eau et de la biodiversité. Ce nouveau dispositif mobilise Santé publique France, chargé de produire et d'interpréter un indicateur de suivi des résultats, et l'Anses, chargée d'harmoniser les méthodes utilisées pour garantir la qualité des analyses.
Dans l'attente du déploiement de ce nouveau réseau, Obépine a bénéficié d'un prolongement de son financement pour poursuivre son activité de surveillance jusqu'en avril 2022328(*), soit avant la mise en fonctionnement de SUM'Eau, ce qui a entraîné une interruption dans la surveillance des eaux usées. Selon Santé publique France, ce délai de mise en oeuvre s'explique par la phase d'appropriation nécessaire et les multiples étapes requises pour rendre ce nouveau réseau opérationnel (définition d'une stratégie d'échantillonnage, validation des méthodes d'analyses et de traitement de données, identification et sélection des laboratoires d'analyse, construction, validation et production automatisée d'indicateurs de suivi, acquisition de l'expertise nécessaire, etc.).
Depuis octobre 2023, les données de surveillance issues du système SUM'Eau, qui repose sur 12 stations de traitement des eaux usées, sont accessibles sur le site data.gouv.fr et intégrées au bulletin hebdomadaire de suivi des infections respiratoires aiguës. Les premiers mois d'exploitation ont révélé une bonne corrélation entre l'indicateur établi et les résultats des tests biologiques ou les passages aux urgences mais n'ont pas montré de précocité significative concernant les changements de tendances. Une phase d'expansion est prévue en 2024 pour atteindre 54 stations surveillées, avec un objectif final de 126 stations. Le réseau SUM'Eau ne permet pas actuellement de séquencer le matériel génétique trouvé dans les eaux usées, mais des travaux exploratoires sont en cours en ce sens. Il est également envisagé d'étendre, à terme, la surveillance à d'autres agents pathogènes tels que la grippe, le VRS ou le poliovirus, sous réserve d'un financement pérenne.
En parallèle de ce nouveau réseau, Obépine poursuit ses activités de recherche, notamment au travers d'un projet collaboratif, dénommé SISP&EAU, financé par l'ANRS-MIE, visant à croiser les données des cas cliniques et celles issues des eaux usées afin d'améliorer la surveillance des virus respiratoires (influenza, VRS, etc.)329(*). Un projet Obépine+, ayant pour objectif de construire une plateforme de recherche et d'innovation destinée à soutenir les réseaux de surveillance à partir des eaux usées en développant, validant et assurant le transfert rapide de méthodes performantes, a été déposé à l'ANR - sur sollicitation du Secrétariat général pour l'investissement - et est actuellement en attente de financement. Afin de construire des liens forts et d'assurer des échanges réguliers permettant une articulation adaptée de la recherche et de la surveillance - qui ont pu faire défaut lors de la construction du réseau SUM'Eau, empêchant de capitaliser pleinement sur l'expérience du réseau Obépine -, il est prévu que les acteurs de SUM'Eau siègent au Comité stratégique d'Obépine+.
L'intérêt pour l'épidémiologie des eaux usées pendant la crise de la covid-19 a permis d'accumuler un corpus de connaissances important et de développer des méthodes offrant de nombreuses perspectives pour la surveillance des maladies humaines ou animales via cette approche330(*). En plus des maladies infectieuses émergentes, la surveillance des eaux usées pourrait à l'avenir être élargie à de nombreux autres champs pour mieux appréhender la santé des populations : suivi de marqueurs biologiques, des usages pharmaceutiques, de l'exposition aux pesticides ou aux produits chimiques, etc.331(*)
* 296 La précocité du suivi dépend toutefois du délai entre l'infection et le début de l'excrétion virale, qui peut différer selon les virus, et de la fréquence et de la rapidité de traitement des analyses des eaux usées.
* 297 Le suivi des eaux usées dans les stations d'épuration ne donne des informations que pour un bassin relativement large de population. Toutefois, si les eaux usées sont prélevées et analysées plus en amont dans le réseau, il est possible de suivre la population plus finement, à l'échelle d'un quartier ou même d'un bâtiment.
* 298 H. Asghar et al., J. Infect. Dis. 2014, 210, S294 ( https://doi.org/10.1093/infdis/jiu384).
* 299 UK Health Security Agency, Poliovirus detected in sewage from North and East London, 2022 ( https://www.gov.uk/government/news/poliovirus-detected-in-sewage-from-north-and-east-london).
* 300 A. K. Debes et al., Am. J. Trop. Med. Hyg.2016, 94, 537 ( https://doi.org/10.4269/ajtmh.15-0496 ).
* 301 P. Callaghan et al., J. Hyg. 1968, 66, 489 ( https://doi.org/10.1017/S0022172400028230).
* 302 a) M. Bisseux et al., Euro. Surveill. 2018, 23, 17 ( https://doi.org/10.2807/1560-7917.es.2018.23.7.17-00237) ; b) C. McCall et al., Water Res. 2020, 184, 116160 ( https://doi.org/10.1016/j.watres.2020.116160).
* 303 Observatoire européen des drogues et des toxicomanies, Wastewater analysis and drugs -- a European multi-city study ( https://www.emcdda.europa.eu/publications/html/pods/waste-water-analysis_en).
* 304 C. D. Metcalfe et al., Environ. Toxicol. Chem. 2010, 29, 79 ( https://doi.org/10.1002/etc.27).
* 305 L'excrétion virale précède l'apparition des symptômes. Voir : V. Maréchal et al., Bull. Acad. Vét. France 2021, 174, 137 ( https://doi.org/10.3406/bavf.2021.70970).
* 306 G. Medema et al., Environ. Sci. Technol. Lett. 2020, 7, 511 ( https://doi.org/10.1021/acs.estlett.0c00357).
* 307 S. Wurtzer et al., Euro. Surveill. 2020, 25, 2000776 ( https://doi.org/10.2807/1560-7917.es.2020.25.50.2000776).
* 308 N. Cluzel et al., Environ. Int. 2022, 158, 106998 ( https://doi.org/10.1016/j.envint.2021.106998).
* 309 C. C. Naughton et al., FEMS Microbes, 2023, 4, 1 ( https://doi.org/10.1093/femsmc/xtad003).
* 310 a) S. Harris-Lovett et al., J. Public Health Manag. Pract. 2023, 29, 317 ( https://doi.org/10.1097/PHH.0000000000001636) ; b) R. R. Spurbeck et al., Sci. Total Environ. 2021, 789, 147829 ( https://doi.org/10.1016/j.scitotenv.2021.147829).
* 311 W.-y. Ng et al., Sci. Total Environ. 2023, 875, 162661 ( https://doi.org/10.1016/j.scitotenv.2023.162661).
* 312 S. W. Olesen et al., Water Res. 2021, 202, 117433 ( https://doi.org/10.1016/j.watres.2021.117433).
* 313 R. Schill et al., Sci. Total Environ. 2023, 871, 162069 ( https://doi.org/10.1016/j.scitotenv.2023.162069).
* 314 K. Bibby et al., Water Res. 2021, 202, 117438 ( https://doi.org/10.1016/j.watres.2021.117438).
*
315 A. Xiao et al., Water Res.
2022, 212, 118070 (
https://doi.org/10.1016/j.watres.2022.118070).
De
la même manière, dans le Nevada, alors qu'aucune avance n'avait
été constatée avec le variant Delta, celle-ci a
été significative au cours de la vague Omicron, probablement en
raison d'une surcharge des capacités de dépistage. Voir : L.
Li et al., Heliyon 2024, 10, e29462 (
https://doi.org/10.1016/j.heliyon.2024.e29462).
* 316 R. Schill et al., Sci. Total Environ. 2023, 871, 162069 ( https://doi.org/10.1016/j.scitotenv.2023.162069).
* 317 Pour s'affranchir de cette contrainte, il est nécessaire de normaliser les résultats avec les débits des sites d'échantillonnage et des biomarqueurs humains permettant d'identifier l'échantillon de population concerné. Voir : D. Polo et al., Water Res. 2020, 186, 116404 ( https://doi.org/10.1016/j.watres.2020.116404).
* 318 D. L. Wannigama et al., Lancet. Infect. Dis. 2024, sous presse ( https://doi.org/10.1016/S1473-3099(24)00155-5).
* 319 a) « Veolia, le CNRS et Université Côte d'Azur traquent les variants du SARS-CoV-2 dans les eaux usées », 2021 ( https://www.cote-azur.cnrs.fr/fr/cnrsinfo/veolia-le-cnrs-et-universite-cote-dazur-traquent-les-variants-du-sars-cov-2-dans-les-eaux) b) G. Rios et al., Lancet Reg. Health Eur. 2021, 10, 100202. ( https://doi.org/10.1016/j.lanepe.2021.100202).
Voir également : a) S. Wurtzer et al., Sci. Total. Environ. 2022, 810, 152213 ( https://doi.org/10.1016/j.scitotenv.2021.152213) ; b) S. Wurtzer et al., Sci. Total. Environ. 2022, 848, 157740 ( https://doi.org/10.1016/j.scitotenv.2022.157740).
* 320 a) D. S. Smyth et al., Nat. Commun. 2022, 13, 635 ( https://doi.org/10.1038/s41467-022-28246-3) ; b) A. C. Reis et al., Sci. Total Environ. 2024, 921, 170961( https://doi.org/10.1016/j.scitotenv.2024.170961).
* 321 F. S. Brunner et al., Water Res. 2023, 247, 120804 ( https://doi.org/10.1016/j.watres.2023.120804).
* 322 a) F. Amman et al., Nat. Biotechnol. 2022, 40, 1814 ( https://doi.org/10.1038/s41587-022-01387-y) ; b) M. Yousif et al.,. Nat. Commun. 2023, 14, 6325 ( https://doi.org/10.1038/s41467-023-41369-5).
* 323 V. Maréchal et al., Bull. Acad. Vét. France 2021, 174, 137 ( https://doi.org/10.3406/bavf.2021.70970).
* 324 La disponibilité des tests reste toutefois nécessaire à l'échelle individuelle dans une perspective de diagnostic.
* 325 On peut également souligner d'autres initiatives de moins grande ampleur comme l'unité Comete (Covid-19 Marseille Environment Testing & Expertise) mise en place par les marins-pompiers de Marseille et le CNRS.
* 326 Voir : https://www.reseau-obepine.fr/carte-des-tendances/.
Des rapports d'analyses fournissant une présentation plus détaillée des résultats par station étaient également publiés. Voir : https://www.reseau-obepine.fr/donnees-ouvertes/.
* 327 Direction générale de l'environnement de la Commission européenne, Recommandation (UE) 2021/472 de la Commission du 17 mars 2021 concernant une approche commune pour la mise en place d'une surveillance systématique de la présence du SARS-CoV-2 et de ses variants dans les eaux usées de l'Union européenne, 2021 ( https://op.europa.eu/fr/publication-detail/-/publication/05b46cb0-8855-11eb-ac4c-01aa75ed71a1/language-fr).
* 328 Un suivi de 30 à 50 stations est resté toutefois en place pour répondre aux activités de recherche.
* 329 Des travaux menés par Obépine avaient déjà montré la possibilité de suivre précocement la circulation du virus Mpox à Paris à partir des eaux usées. Voir : S. Wurtzer et al., Environ. Sci. Technol. Lett. 2022, 9, 991 ( https://doi.org/10.1021/acs.estlett.2c00693).
* 330 V. Maréchal et al., Anaesth. Crit. Care Pain Med. 2023, 42, 101251
( https://doi.org/10.1016/j.accpm.2023.101251).
* 331 P. M. Choi et al., Trends Anal. Chem. 2018, 105, 453 ( https://doi.org/10.1016/j.trac.2018.06.004).