E. LA SURVEILLANCE GÉNOMIQUE
S'il existait des doutes quant à la capacité d'adaptation du SARS-CoV-2 au début de la pandémie, le virus a rapidement montré sa capacité à évoluer génétiquement, avec des conséquences sur sa transmissibilité, sur les populations touchées, la gravité de la maladie et sur l'efficacité des thérapies, des vaccins et des tests diagnostiques332(*). Le suivi de l'évolution génomique du virus revêt une importance cruciale, au même titre que la surveillance de la dynamique épidémique. Ce suivi permet de détecter l'émergence et la propagation des variants d'intérêt, susceptibles d'entraîner une recrudescence d'infections, et d'étudier leurs caractéristiques (transmissibilité, pathogénicité, etc.) afin de prendre les mesures appropriées pour limiter leur diffusion et leurs impacts en termes de santé. C'est sur la base de cette surveillance génomique que les vaccins contre la covid-19 ont pu être adaptés aux souches circulantes du virus, pour une meilleure protection de la population. Au regard de l'importance de cette surveillance génomique, la Commission européenne a appelé dès janvier 2021 - soit peu après la propagation du variant Alpha en fin d'année 2020 - les États membres à développer une capacité de séquençage d'au moins 5 % - et de préférence 10 % - des échantillons de dépistage positifs333(*).
Au début de la crise, la France accusait un certain retard en la matière, avec moins de 3 000 séquences publiées en 2020, contre plus de 140 000 par le Royaume-Uni334(*). Aussi, en janvier 2021, Santé publique France et l'ANRS-MIE ont lancé le consortium Emergen (surveillance et recherche sur les infections à pathogènes émergents via la génomique microbienne), à l'instar du consortium britannique COG-UK, afin d'identifier l'émergence de nouveaux variants et les proportions des différents variants circulant sur le territoire français. Dans ce cadre, un réseau de collecte d'échantillons a été établi, en s'appuyant sur des laboratoires de ville et hospitaliers, les capacités de séquençage ont été augmentées, grâce à un investissement matériel et au recours à des laboratoires privés, et une plateforme informatique dédiée, permettant de collecter, traiter et contrôler l'ensemble des données recueillies, a été développée. Afin de rendre disponibles les résultats obtenus pour la surveillance sanitaire et la recherche, l'ensemble des séquences étaient notamment déposées dans les entrepôts de données internationaux, tels que GISAID (Global Initiative on Sharing All Influenza Data). Grâce à ce consortium, l'activité de séquençage française a été considérablement multipliée, passant de 3 000 séquences en 2020 à 300 000 en 2021, pour un total de 750 000 séquences génomiques virales produites de janvier 2021 à juin 2023, permettant à la France de se placer au quatrième rang mondial des contributeurs à GISAID.
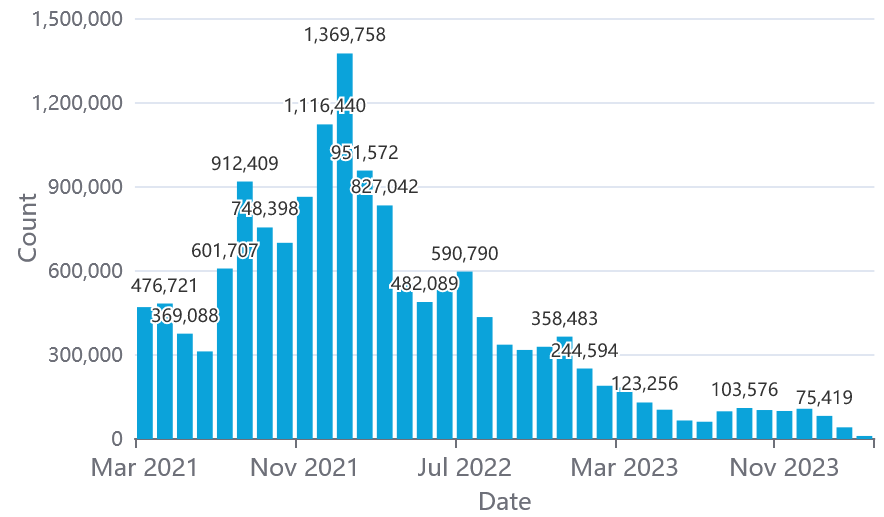
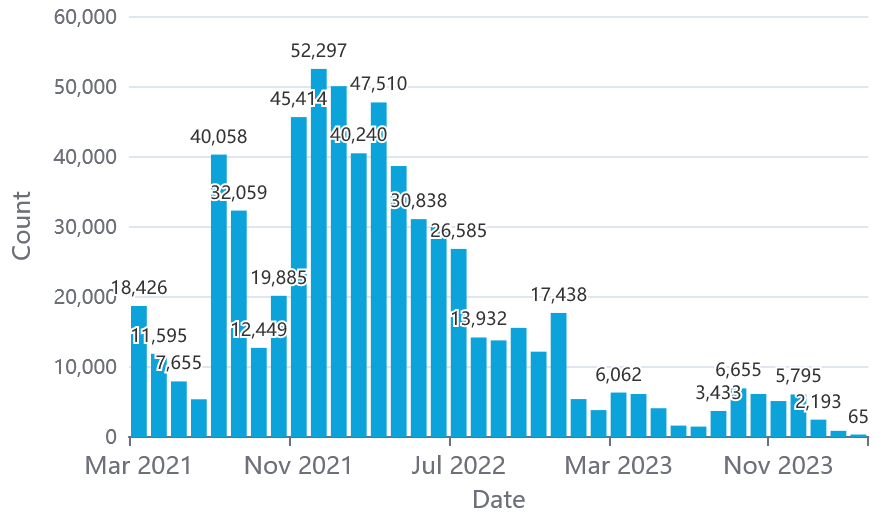
L'évolution de la pandémie nécessitant moins de données, le consortium a évolué et ajusté ses activités en début d'année 2023, poursuivant l'effort de séquençage à un rythme plus modéré à partir des seules ressources du Centre national de référence (CNR) des Virus des infections respiratoires et de ses laboratoires associés. L'évolution de la stratégie de dépistage et le plus faible recours actuel aux tests ont un impact sur la surveillance génomique, en France comme dans le reste du monde, comme le montre l'évolution du nombre de séquences déposées sur GISAID (voir figure ci-après). Pour répondre à cette situation, de nouveaux laboratoires ont été récemment mobilisés pour la collecte d'échantillons biologiques positifs, en complément du réseau déjà actif. Selon Santé publique France, cette diminution ne représentait pas, lors de son audition, un obstacle à une bonne surveillance des variants en raison d'un nombre de séquences - entre 100 et 500 par semaine - qui restait suffisant pour disposer d'une vision satisfaisante des souches circulantes et de leurs tendances. Toutefois, dans un contexte de faible circulation du SARS-CoV-2, on peut noter une récente baisse du nombre de séquences disponibles, parfois inférieur à 100 par semaine, depuis mars 2024335(*).
Nombre de séquences déposées sur GISAID dans le monde (gauche) et en France (droite)336(*)
Le projet Emergen se prépare désormais à une nouvelle étape au travers de deux initiatives : le projet Emergen 2.0, en attente de financement par l'ANR, et le projet SEQ4EPI337(*), qui bénéficie du soutien financier de l'Union européenne via le programme EU4Health. Ces projets visent à consolider et élargir le système établi en soutenant la surveillance d'autres maladies infectieuses et en élargissant le suivi à de nouveaux agents pathogènes pouvant représenter des menaces pandémiques potentielles. L'augmentation des capacités résultant de la structuration du consortium Emergen a déjà permis au CNR des Virus des infections respiratoires de se fixer des objectifs plus ambitieux pour la surveillance génomique des virus influenza et de démarrer le séquençage du VRS. Dans la perspective d'une approche « une seule santé », il est également prévu d'élargir les analyses à la surveillance animale pour identifier les risques de futures zoonoses, en collaboration avec l'Anses.
* 332 Voir « Le suivi des variants du SARS-CoV-2 » dans le rapport fait au nom de l'Office parlementaire d'évaluation des choix scientifiques et technologiques sur « les aspects scientifiques et techniques de la lutte contre la pandémie de la covid-19 » par MM. Jean-François Eliaou et Gérard Leseul, députés, et Mmes Sonia de La Provôté et Florence Lassarade, sénatrices ( http://www.senat.fr/notice-rapport/2020/r20-741-notice.html) et « Quelles perspectives pour la pandémie de covid-19 ? Faut-il faire évoluer la stratégie de lutte contre le virus ? » dans le rapport fait au nom de l'Office parlementaire d'évaluation des choix scientifiques et technologiques sur « la lutte contre la pandémie de la covid-19 : aspects scientifiques et techniques - conséquences indirectes » par MM. Jean-François Eliaou et Gérard Leseul, députés, et Mmes Sonia de La Provôté et Florence Lassarade, sénatrices ( https://www.senat.fr/rap/r21-531/r21-5311.pdf).
* 333 Commission européenne, Communication de la Commission au Parlement européen, au Conseil européen, et au Conseil : Un front uni pour vaincre la covid-19, 2021 ( https://eur-lex.europa.eu/legal-content/EN/ALL/?uri=COM%3A2021%3A35%3AFIN).
* 334 H. Touzet et al., Le SARS-CoV-2 : une expérience inédite de surveillance génomique mondiale, 2023 ( https://inria.hal.science/hal-04155812/).
* 335 Activités de séquençage au sein du consortium Emergen ( https://emergen-db.france-bioinformatique.fr/fr/external/report/dashboard/).
* 336 Voir : https://gisaid.org/hcov-19-variants-dashboard/.
* 337 Voir : https://www.santepubliquefrance.fr/dossiers/coronavirus-covid-19/projet-seq4epi.